若有任何問題請來信:gss_crm@gss.com.tw
離群值(Outlier)顧名思義即為某些數據與其它多數的數據有著明顯不同的分佈情況,然而離群值的發生可能有很多原因,例如:人為疏失、硬體故障、惡意行為等,故當離群值發生時不可輕忽,以下簡單介紹三種方法,用以檢測資料是否有離群值的發生。
方法1:箱形圖
箱形圖(Box plot)又稱為盒鬚圖,是種方法簡單並且可快速觀察出資料的簡易分配狀況的圖形,且其最大的優點是該種方法並不要求資料屬於何種分配,屬於無母數方法,故被廣泛使用。
其找出離群值的方法為:
1.將資料進行排序後,找出第一四分位數(Q1)、第三四分位數(Q3)。
2.計算 Q3 - Q1 得出四分位距(IQR)。
3.找出數據中大於 Q3 +1.5IQR 或 小於Q1 -1.5IQR的數據, 即為離群值。
以下透過R來進行實踐:
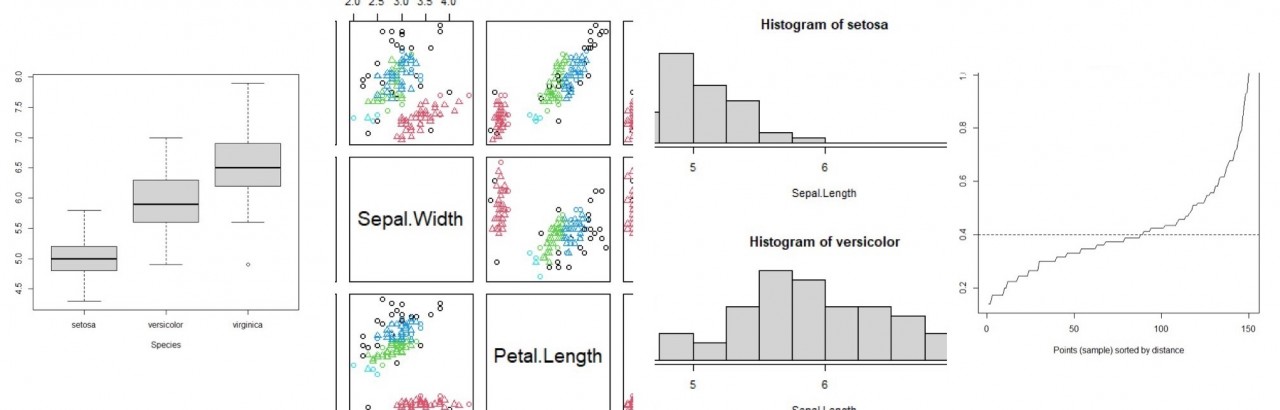
以iris的資料集為例,該資料集紀錄3種品種的鳶蕊花資料,此處我們僅先使用Species(品種)、Sepal.Length(花顎長度)進行分析。
iris_data <- iris
box_info <- boxplot(Sepal.Length ~ Species ,data = iris_data)
> box_info$group
[1] 3
> box_info$names
[1] "setosa" "versicolor" "virginica"
> box_info$out
[1] 4.9
> virginica_summary <- box_info$stats[,3]
> names(virginica_summary) <- c("Min","Q1","Q2","Q3","Max")
> virginica_summary
Min Q1 Q2 Q3 Max
5.6 6.2 6.5 6.9 7.9
透過箱形圖可知,在品種setosa以及versicolor並沒有離群值的產生,然而在virginica存在一筆花顎長度為4.9的離群值(以空心圓表示),此品種的最小值、Q1、Q2、Q3、最大值依序為5.6、6.2、6.5、6.9及7.9,該離群值明顯與其他的數據有明顯偏離。
方法2.Z分數法
當某一隨機變數服從常態分布(normal distribution)時,該隨機變數之平均值正負1、2、3個標準差的區間之面積,佔總面積分別約68%、95%、99.7%,此處先繪製直方圖觀察各品種的分布情形,
breaks <- seq(4, 8, length.out = 17) par(mfrow=c(3,1)) hist(subset(iris_data, Species == "setosa" , select = c(Sepal.Length))$Sepal.Length,xlim= c(4,8),xlab = "Sepal.Length",main = "Histogram of setosa", breaks = breaks) hist(subset(iris_data, Species == "versicolor" , select = c(Sepal.Length))$Sepal.Length,xlim= c(4,8),xlab = "Sepal.Length",main = "Histogram of versicolor",breaks = breaks) hist(subset(iris_data, Species == "virginica" , select = c(Sepal.Length))$Sepal.Length,xlim= c(4,8),xlab = "Sepal.Length",main = "Histogram of virginica" ,breaks = breaks)
由上圖觀察可知,此三類品種的分部大致呈現兩側較低中間較高的情況,接著我們透過Shapiro-Wilk 常態性檢定,判斷此三類品種在花顎長度是否為常態分配。
> shapiro.test(subset(iris_data, Species == "setosa" , select = c(Sepal.Length))$Sepal.Length)
Shapiro-Wilk normality test
data: subset(iris_data, Species == "setosa", select = c(Sepal.Length))$Sepal.Length
W = 0.9777, p-value = 0.4595
> shapiro.test(subset(iris_data, Species == "versicolor" , select = c(Sepal.Length))$Sepal.Length)
Shapiro-Wilk normality test
data: subset(iris_data, Species == "versicolor", select = c(Sepal.Length))$Sepal.Length
W = 0.97784, p-value = 0.4647
> shapiro.test(subset(iris_data, Species == "virginica" , select = c(Sepal.Length))$Sepal.Length)
Shapiro-Wilk normality test
data: subset(iris_data, Species == "virginica", select = c(Sepal.Length))$Sepal.Length
W = 0.97118, p-value = 0.2583
檢定的結果顯示,三類的p-value皆大於0.05,故不拒虛無假設(H:花顎長度符合常態分布),皆著我們對三類品種的花顎長度各別進行標準化,即各數值減去該組平均值再除以標準差,使各組之平均值為、標準差為1:
> iris_data$zscore <- ave(iris_data$Sepal.Length, iris_data$Species, FUN=scale)
> head(iris_data[,c("Sepal.Length","Species","zscore")])
Sepal.Length Species zscore
1 5.1 setosa 0.26667447
2 4.9 setosa -0.30071802
3 4.7 setosa -0.86811050
4 4.6 setosa -1.15180675
5 5.0 setosa -0.01702177
6 5.4 setosa 1.11776320
> subset(iris_data,zscore > 3 | zscore < -3 )
[1] Sepal.Length Sepal.Width Petal.Length Petal.Width Species zscore
<0 rows> (or 0-length row.names)
其中zscore即為標準化後的欄位,當該數值大於3或小於3時,該組數據即可視為離群值,但結果顯示,並沒有任何一組數據被此方法視為離群值。
補充1:若隨機變數符合常態分部時,在方法1中,介於Q1 -1.5IQR 與Q3 +1.5IQR 之間的面積佔總體面積約為99.3%。
補充2:醫療的品質管制有時會透過4種管制圖進行監測控管,包含:p-chart、np-chart、c-chart、u-chart,其概念與z分數法大致相同,皆是透過檢測數據是否有正常落在平均值加減3個標準差之內進行控管,最大的差別僅在於對資料個數或資料的分配的假設有所不同而已。
方法3:DBSCAN
DBSCAN(Density-based spatial clustering of applications with noise),是一種分群的非監督式演算法,其基礎概念,某一點p在一定的距離ε內,至少有MinPts個數據時,即可將其分成一群,換句話說就是鄰近的數據集是否滿足一定的密度,當滿足時則可進行分群(詳細步驟可參考下方來源)。故當某些數據點遠離其他數據時,則有可能無法被分類成任意一群時,此時即視為離群值。
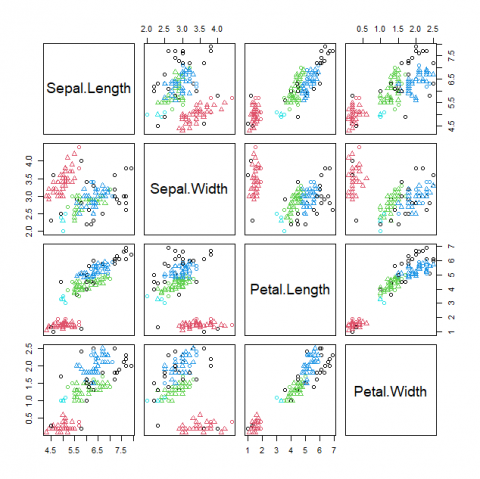
此次改為使用鳶蕊花資料集的Sepal length(花萼長度)、Sepal width(花萼寬度)、Petal length(花瓣長度)、Petal width(花瓣寬度)欄位進行分群並找出離群值,此次使用的DBSCAN 之套件為fpc。
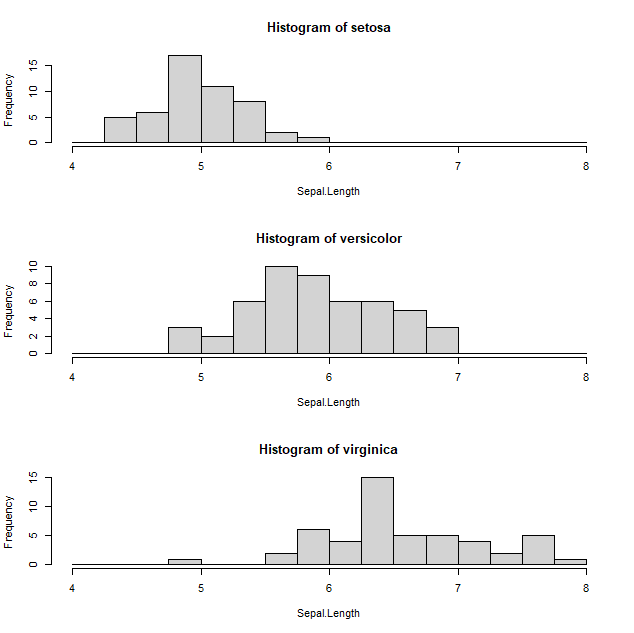
此處我們先設MinPts = 4 ,而eps則可透過函式kNNdist進行判斷。
> library(fpc) > dbscan::kNNdistplot(iris_data[,1:4], k = 4) > abline(h = 0.4, lty = 2)
圖中我們找出轉折處約為0.4作為dbscan的eps參數並開始分群。
> dbscan.result <- dbscan(iris_data[,1:4],eps=0.4,MinPts=4)
> table(iris_data$Species, dbscan.result$cluster)
0 1 2 3 4
setosa 3 47 0 0 0
versicolor 5 0 38 3 4
virginica 17 0 0 33 0
> plot(dbscan.result,iris_data[,1:4])
結果DBSCAN共將資料分成了四群以及另有25筆數據被視為離群值,我們可以透過混淆矩陣進行觀察,其中setosa、versicolor、virginica分別有3、5、17筆資料被視為離群值,且大致被依序分到第1、2、3群,versicolor有少數幾筆資料被分到第3、4群。另外我們從二維圖形可觀察到,其中黑色的點即為離群值,確實大多被分在較邊緣或與呈現密度較低的位置。
DBSCAN不但可以幫助資料進行分群,同時可以找出離群值,但同時也有一些缺點,即在不同的情況下,有時可能無法選出較好的eps以及MinPts,使其有較好的分類結果,例如:維度災難、 資料分布密度不同等。
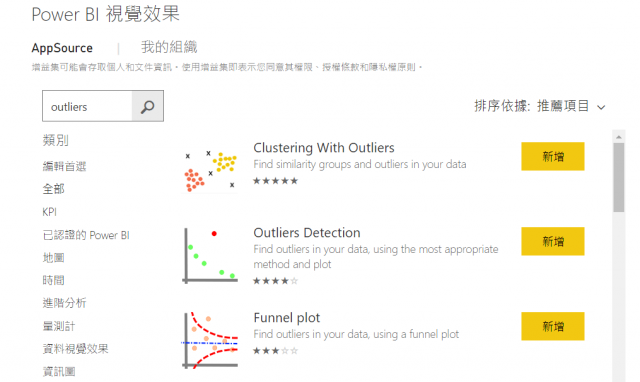
如果對於R還不熟悉的話,也可以嘗試透過微軟的Power BI找出離群值,在AppSource也提供許多種不同的視覺化效果可進行分析,例如:Outliers Detection、Clustering With Outliers、Funnel plot,特別的是,此三個視覺化效果也有使用R的Packages進行實踐的。其中Outliers Detection就包含了前述的箱形圖以及Z分數法,而Clustering With Outliers則包含了DBSCAN之演算法。
結論:
本次僅簡介三種關於找出離群值的方法,如果在進行資料分析或應用時,無視於資料內的離群值的存在,可能會導致分析結果較難以解釋,亦或是出現偏差,但也不可因此任意將離群值刪除,應審慎評估為何有離群值的產生,或是資料分析的目的性等,再審慎的處理之。
參考來源:
1.WIKI:DBSCAN
2.dbscan - Density Based Clustering of Applications with Noise (DBSCAN) and Related Algorithms - R package
3.【機器學習】基於密度的聚類演算法 DBSCAN
4.極端值判斷教學